Proteogenomic Workflow Reveals Molecular Phenotypes Related to Breast Cancer Mammographic Appearance
- PMID: 33855848
- PMCID: PMC8155562
- DOI: 10.1021/acs.jproteome.1c00243
Proteogenomic Workflow Reveals Molecular Phenotypes Related to Breast Cancer Mammographic Appearance
Abstract
Proteogenomic approaches have enabled the generat̲ion of novel information levels when compared to single omics studies although burdened by extensive experimental efforts. Here, we improved a data-independent acquisition mass spectrometry proteogenomic workflow to reveal distinct molecular features related to mammographic appearances in breast cancer. Our results reveal splicing processes detectable at the protein level and highlight quantitation and pathway complementarity between RNA and protein data. Furthermore, we confirm previously detected enrichments of molecular pathways associated with estrogen receptor-dependent activity and provide novel evidence of epithelial-to-mesenchymal activity in mammography-detected spiculated tumors. Several transcript-protein pairs displayed radically different abundances depending on the overall clinical properties of the tumor. These results demonstrate that there are differentially regulated protein networks in clinically relevant tumor subgroups, which in turn alter both cancer biology and the abundance of biomarker candidates and drug targets.
Keywords: breast cancer; data-independent acquisition; proteogenomics; proteomics; transcriptomics.
Conflict of interest statement
The authors declare no competing financial interest.
Figures
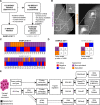


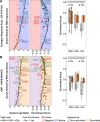


References
-
- Fachal L.; Aschard H.; Beesley J.; Barnes D. R.; Allen J.; Kar S.; Pooley K. A.; Dennis J.; Michailidou K.; Turman C.; Soucy P.; Lemaçon A.; Lush M.; Tyrer J. P.; Ghoussaini M.; Marjaneh M. M.; Jiang X.; Agata S.; Aittomäki K.; Alonso M. R.; Andrulis I. L.; Anton-Culver H.; Antonenkova N. N.; Arason A.; Arndt V.; Aronson K. J.; Arun B. K.; Auber B.; Auer P. L.; Azzollini J.; Balmaña J.; Barkardottir R. B.; Barrowdale D.; Beeghly-Fadiel A.; Benitez J.; Bermisheva M.; Białkowska K.; Blanco A. M.; Blomqvist C.; Blot W.; Bogdanova N. V.; Bojesen S. E.; Bolla M. K.; Bonanni B.; Borg A.; Bosse K.; Brauch H.; Brenner H.; Briceno I.; Brock I. W.; Brooks-Wilson A.; Brüning T.; Burwinkel B.; Buys S. S.; Cai Q.; Caldés T.; Caligo M. A.; Camp N. J.; Campbell I.; Canzian F.; Carroll J. S.; Carter B. D.; Castelao J. E.; Chiquette J.; Christiansen H.; Chung W. K.; Claes K. B. M.; Clarke C. L.; Collée J. M.; Cornelissen S.; Couch F. J.; Cox A.; Cross S. S.; Cybulski C.; Czene K.; Daly M. B.; de la Hoya M.; Devilee P.; Diez O.; Ding Y. C.; Dite G. S.; Domchek S. M.; Dörk T.; Dos-Santos-Silva I.; Droit A.; Dubois S.; Dumont M.; Duran M.; Durcan L.; Dwek M.; Eccles D. M.; Engel C.; Eriksson M.; Evans D. G.; Fasching P. A.; Fletcher O.; Floris G.; Flyger H.; Foretova L.; Foulkes W. D.; Friedman E.; Fritschi L.; Frost D.; Gabrielson M.; Gago-Dominguez M.; Gambino G.; Ganz P. A.; Gapstur S. M.; Garber J.; García-Sáenz J. A.; Gaudet M. M.; Georgoulias V.; Giles G. G.; Glendon G.; Godwin A. K.; Goldberg M. S.; Goldgar D. E.; González-Neira A.; Tibiletti M. G.; Greene M. H.; Grip M.; Gronwald J.; Grundy A.; Guénel P.; Hahnen E.; Haiman C. A.; Håkansson N.; Hall P.; Hamann U.; Harrington P. A.; Hartikainen J. M.; Hartman M.; He W.; Healey C. S.; Heemskerk-Gerritsen B. A. M.; Heyworth J.; Hillemanns P.; Hogervorst F. B. L.; Hollestelle A.; Hooning M. J.; Hopper J. L.; Howell A.; Huang G.; Hulick P. J.; Imyanitov E. N.; Isaacs C.; Iwasaki M.; Jager A.; Jakimovska M.; Jakubowska A.; James P. A.; Janavicius R.; Jankowitz R. C.; John E. M.; Johnson N.; Jones M. E.; Jukkola-Vuorinen A.; Jung A.; Kaaks R.; Kang D.; Kapoor P. M.; Karlan B. Y.; Keeman R.; Kerin M. J.; Khusnutdinova E.; Kiiski J. I.; Kirk J.; Kitahara C. M.; Ko Y.-D.; Konstantopoulou I.; Kosma V.-M.; Koutros S.; Kubelka-Sabit K.; Kwong A.; Kyriacou K.; Laitman Y.; Lambrechts D.; Lee E.; Leslie G.; Lester J.; Lesueur F.; Lindblom A.; Lo W.-Y.; Long J.; Lophatananon A.; Loud J. T.; Lubiński J.; MacInnis R. J.; Maishman T.; Makalic E.; Mannermaa A.; Manoochehri M.; Manoukian S.; Margolin S.; Martinez M. E.; Matsuo K.; Maurer T.; Mavroudis D.; Mayes R.; McGuffog L.; McLean C.; Mebirouk N.; Meindl A.; Miller A.; Miller N.; Montagna M.; Moreno F.; Muir K.; Mulligan A. M.; Muñoz-Garzon V. M.; Muranen T. A.; Narod S. A.; Nassir R.; Nathanson K. L.; Neuhausen S. L.; Nevanlinna H.; Neven P.; Nielsen F. C.; Nikitina-Zake L.; Norman A.; Offit K.; Olah E.; Olopade O. I.; Olsson H.; Orr N.; Osorio A.; Pankratz V. S.; Papp J.; Park S. K.; Park-Simon T.-W.; Parsons M. T.; Paul J.; Pedersen I. S.; Peissel B.; Peshkin B.; Peterlongo P.; Peto J.; Plaseska-Karanfilska D.; Prajzendanc K.; Prentice R.; Presneau N.; Prokofyeva D.; Pujana M. A.; Pylkäs K.; Radice P.; Ramus S. J.; Rantala J.; Rau-Murthy R.; Rennert G.; Risch H. A.; Robson M.; Romero A.; Rossing M.; Saloustros E.; Sánchez-Herrero E.; Sandler D. P.; Santamariña M.; Saunders C.; Sawyer E. J.; Scheuner M. T.; Schmidt D. F.; Schmutzler R. K.; Schneeweiss A.; Schoemaker M. J.; Schöttker B.; Schürmann P.; Scott C.; Scott R. J.; Senter L.; Seynaeve C. M.; Shah M.; Sharma P.; Shen C.-Y.; Shu X.-O.; Singer C. F.; Slavin T. P.; Smichkoska S.; Southey M. C.; Spinelli J. J.; Spurdle A. B.; Stone J.; Stoppa-Lyonnet D.; Sutter C.; Swerdlow A. J.; Tamimi R. M.; Tan Y. Y.; Tapper W. J.; Taylor J. A.; Teixeira M. R.; Tengström M.; Teo S. H.; Terry M. B.; Teulé A.; Thomassen M.; Thull D. L.; Tischkowitz M.; Toland A. E.; Tollenaar R. A. E. M.; Tomlinson I.; Torres D.; Torres-Mejía G.; Troester M. A.; Truong T.; Tung N.; Tzardi M.; Ulmer H.-U.; Vachon C. M.; van Asperen C. J.; van der Kolk L. E.; van Rensburg E. J.; Vega A.; Viel A.; Vijai J.; Vogel M. J.; Wang Q.; Wappenschmidt B.; Weinberg C. R.; Weitzel J. N.; Wendt C.; Wildiers H.; Winqvist R.; Wolk A.; Wu A. H.; Yannoukakos D.; Zhang Y.; Zheng W.; Hunter D.; Pharoah P. D. P.; Chang-Claude J.; García-Closas M.; Schmidt M. K.; Milne R. L.; Kristensen V. N.; French J. D.; Edwards S. L.; Antoniou A. C.; Chenevix-Trench G.; Simard J.; Easton D. F.; Kraft P.; Dunning A. M. Fine-Mapping of 150 Breast Cancer Risk Regions Identifies 191 Likely Target Genes. Nat. Genet. 2020, 52, 56–73. 10.1038/s41588-019-0537-1. - DOI - PMC - PubMed
-
- Perou C. M.; Sørlie T.; Eisen M. B.; van de Rijn M.; Jeffrey S. S.; Rees C. a.; Pollack J. R.; Ross D. T.; Johnsen H.; Akslen L. A.; Fluge O.; Pergamenschikov A.; Williams C.; Zhu S. X.; Lønning P. E.; Børresen-Dale A. L.; Brown P. O.; Botstein D. Molecular Portraits of Human Breast Tumours. Nature 2000, 406, 747–752. 10.1038/35021093. - DOI - PubMed
-
- Coates A. S.; Winer E. P.; Goldhirsch A.; Gelber R. D.; Gnant M.; Piccart-Gebhart M.; Thürlimann B.; Senn H.-J. Panel Members. Tailoring Therapies--Improving the Management of Early Breast Cancer: St Gallen International Expert Consensus on the Primary Therapy of Early Breast Cancer 2015. Ann. Oncol. Off. J. Eur. Soc. Med. Oncol. 2015, 26, 1533–1546. 10.1093/annonc/mdv221. - DOI - PMC - PubMed
Publication types
MeSH terms
LinkOut - more resources
Full Text Sources
Other Literature Sources
Medical
Molecular Biology Databases

