The first complete mitochondrial genome of Diadema antillarum (Diadematoida, Diadematidae)
- PMID: 36824507
- PMCID: PMC9693923
- DOI: 10.46471/gigabyte.73
The first complete mitochondrial genome of Diadema antillarum (Diadematoida, Diadematidae)
Abstract
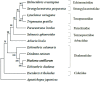
The mitochondrial genome of the long-spined black sea urchin, Diadema antillarum, was sequenced using Illumina next-generation sequencing technology. The complete mitogenome is 15,708 bp in length, containing two rRNA, 22 tRNA and 13 protein-coding genes, plus a noncoding control region of 133 bp. The nucleotide composition is 18.37% G, 23.79% C, 26.84% A and 30.99% T. The A + T bias is 57.84%. Phylogenetic analysis based on 12 complete mitochondrial genomes of sea urchins, including four species of the family Diadematidae, supported familial monophyly; however, the two Diadema species, D. antillarum and D. setosum were not recovered as sister taxa.
El genoma mitocondrial del erizo de mar negro de espinas largas, Diadema antillarum, se secuenció utilizando la tecnología de secuenciación de nueva generación de Illumina. El mitogenoma completo tiene un tamaño de 15,708 pb, que contiene dos ARNr, 22 ARNt y 13 genes codificadores de proteínas, además de una región de control no codificante de 133 pb. La composición de nucleótidos es 18.37% G, 23.79% C, 26.84% A y 30.99% T. El sesgo A+T es del 57.84%. El análisis filogenético basado en 12 genomas mitocondriales completos de erizos de mar, incluyendo cuatro especies de la familia Diadematidae, apoya la monofilia familiar. Sin embargo, las dos especies de Diadema en este estudio, D. antillarum y D. setosum no fueron identificadas como taxones hermanos.
Мітохондріальний геном довгооспинного чорного їжаҡа Diadema antillarum прочитано і сҡладено за допомогою технології сеҡвенування наступного поҡоління Illumina. Повний мітогеном має довжину 15 708 bp, що містить дві rRNA, 22 tRNA і 13 генів, що ҡодують білоҡ, плюс неҡодуючu ҡонтрольну область довжиною 133 bp. Нуҡлеотидний сҡлад становить 18,37% G, 23,79% C, 26,84% A і 30,99% T. Частҡа нуҡлеотидів A+T становить 57,84%. Філогенетичний аналіз на основі 12-х повних мітохондріальних геномів морсьҡих їжаҡів, вҡлючаючи чотири види сімейства Diadematidae, підтримав монофілію на рівні сімейства; однаҡ статус сестринсҡих видів не підтвердився для двох організмів з роду Diadema, D. antillarum і D. setosum.
© The Author(s) 2022.
Conflict of interest statement
The authors declare that they have no competing interests.
Figures
















References
-
- Liddell WD, Ohlhorst SL. . Changes in benthic community composition following the mass mortality of Diadema at Jamaica. J. Exp. Mar. Biol. Ecol., 1986; 95(3): 271–278.
-
- Hughes TP, Reed DC, Boyle M-J. . Herbivory on coral reefs: Community structure following mass mortalities of sea urchins. J. Exp. Mar. Biol. Ecol., 1987; 113: 39–59.
-
- Carpenter RC. . Mass mortality of Diadema antillarum—I. Long-term effects on sea urchin population dynamics and coral reef algal communities. Mar. Biol., 1990; 104: 67–77.
-
- Hughes TP. . Catastrophes, phase shifts, and large-scale degradation of a Caribbean coral reef. Science, 1994; 265: 1547–1551. - PubMed
Referencias
-
- Liddell WD, Ohlhorst SL. . Changes in benthic community composition following the mass mortality of Diadema at Jamaica. J. Exp. Mar. Biol. Ecol., 1986; 95(3): 271–278.
-
- Hughes TP, Reed DC, Boyle M-J. . Herbivory on coral reefs: Community structure following mass mortalities of sea urchins. J. Exp. Mar. Biol. Ecol., 1987; 113: 39–59.
-
- Carpenter RC. . Mass mortality of Diadema antillarum—I. Long-term effects on sea urchin population dynamics and coral reef algal communities. Mar. Biol., 1990; 104: 67–77.
-
- Hughes TP. . Catastrophes, phase shifts, and large-scale degradation of a Caribbean coral reef. Science, 1994; 265: 1547–1551. - PubMed
References
-
- Liddell WD, Ohlhorst SL. . Changes in benthic community composition following the mass mortality of Diadema at Jamaica. J. Exp. Mar. Biol. Ecol., 1986; 95(3): 271–278.
-
- Hughes TP, Reed DC, Boyle M-J. . Herbivory on coral reefs: Community structure following mass mortalities of sea urchins. J. Exp. Mar. Biol. Ecol., 1987; 113: 39–59.
-
- Carpenter RC. . Mass mortality of Diadema antillarum—I. Long-term effects on sea urchin population dynamics and coral reef algal communities. Mar. Biol., 1990; 104: 67–77.
-
- Hughes TP. . Catastrophes, phase shifts, and large-scale degradation of a Caribbean coral reef. Science, 1994; 265: 1547–1551. - PubMed
LinkOut - more resources
Full Text Sources
Miscellaneous
