Prevalence and predictors of NTM in presumed/confirmed drug-resistant TB
- PMID: 39035428
- PMCID: PMC11257093
- DOI: 10.5588/ijtldopen.24.0242
Prevalence and predictors of NTM in presumed/confirmed drug-resistant TB
Abstract
Introduction: Non-tuberculous mycobacteria (NTM) are increasingly isolated in individuals with presumed/confirmed pulmonary TB. We aimed to estimate the prevalence and species distribution of NTM among presumed/confirmed drug-resistant TB (DR-TB) individuals and determine NTM isolation predictors.
Methods: Sputum samples collected for DR-TB diagnosis and follow-up from 2012 to 2021 in Ghana were retrospectively analysed. Samples were subjected to sputum smear microscopy (SSM) and mycobacterial culture. The MPT64 assay was performed on positive cultures to distinguish between Mycobacterium tuberculosis complex MTBc and NTM. NTM isolates were re-cultured for species identification using GenoType® Mycobacterium CM/AS line-probe assay, polymerase chain reaction, and Sanger sequencing targeting 16S rRNA and rpoB genes. MTBc isolates identified by GenoType underwent spoligotyping. A logistic regression model was used to identify the predictors of NTM isolation.
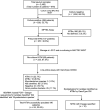
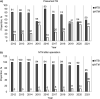
Results: Of the 2,492 samples, 839 (33.7%) tested culture-positive for mycobacteria, with 257 (30.6%) presumed to be NTM. Of these, 53 (23.6%) were identified at the species level, with a predominance of M. intracellulare (66.0%). MPT64 testing missed 18 (3%) MTBc isolates. Logistic regression showed increased odds of NTM isolation in follow-up samples (aOR 2.41, 95% CI 1.46-3.99). NTM species were isolated from 46 patients, with four classified as NTM pulmonary disease.
Conclusion: Enhancing our understanding of local NTM epidemiology and improving local diagnostic capabilities can optimise patient management strategies and outcomes.
Introduction: Les mycobactéries non tuberculeuses (NTM) sont de plus en plus souvent isolées chez les personnes atteintes de TB pulmonaire présumée/confirmée (DR-TB). Notre étude visait à évaluer la fréquence et la répartition des différentes espèces de NTM chez les personnes atteintes de TB pharmacorésistante, ainsi qu'à identifier les facteurs prédictifs de l'isolement de ces NTM.
Méthodes: Les échantillons d'expectorations collectés entre 2012 et 2021 au Ghana pour le diagnostic et le suivi de la DR-TB ont été analysés rétrospectivement. Les échantillons ont subi une microscopie du frottis d'expectoration (SSM) et une culture mycobactérienne. Le test MPT64 a été réalisé sur les cultures positives pour différencier le complexe Mycobacterium tuberculosis (MTBc) et les NTM. Les isolats de NTM ont été soumis à une nouvelle culture pour identification des espèces à l'aide du test par sondes en ligne GenoType® Mycobacterium CM/AS, de l'amplification en chaîne par polymérase et du séquençage de Sanger ciblant les gènes 16S rRNA et rpoB. Les isolats de MTBc identifiés par GenoType ont été soumis à un spoligotypage. Un modèle de régression logistique a été utilisé pour identifier les facteurs prédictifs de l'isolement des NTM.
Résultats: Parmi les 2 492 échantillons analysés, 839 (33,7%) ont été testés positifs à la culture de mycobactéries, dont 257 (30,6%) étaient présumés être des NTM. Parmi ces échantillons, 53 (23,6%) ont été identifiés au niveau de l'espèce, avec une prédominance de M. intracellulare (66,0%). Le test MPT64 a échoué à détecter 18 (3%) isolats de MTBc. L'analyse de régression logistique a révélé une probabilité accrue d'isolement de MNT dans les échantillons de suivi (aOR 2,41 ; IC à 95% 1,46–3,99). Des espèces de NTM ont été isolées chez 46 patients, dont quatre ont été classés dans la catégorie des maladies pulmonaires à NTM.
Conclusion: Une connaissance approfondie de l'épidémiologie locale des NTM et le renforcement des compétences de diagnostic au niveau local peuvent améliorer les stratégies de prise en charge des patients et les résultats obtenus.
Keywords: drug-resistant tuberculosis; non-tuberculous mycobacteria; non-tuberculous mycobacterial pulmonary disease.
© 2024 The Authors.
Conflict of interest statement
Conflicts of interest: none declared.
Figures
References
-
- Cowman S, et al. Non-tuberculous mycobacterial pulmonary disease. Eur Respir J. 2019;54(1):1900250. - PubMed
-
- Aberi R, et al. Magnitude and geographical distribution of non-tuberculous mycobacteria among people presumed to have pulmonary tuberculosis in Kenya. Afr J Health Sci 2021;34(6):696–705.
-
- Haworth CS, et al. British Thoracic Society guidelines for the management of non-tuberculous mycobacterial pulmonary disease (NTM-PD). Vol. 72, Thorax. 2017. 1–64 p. - PubMed
LinkOut - more resources
Full Text Sources
Research Materials
